Was ist Gen-Editing?
Das Gen Editing ist eine Methode zur genetischen Manipulation, mit der Teile der DNA eines Organismus entfernt, hinzugefügt, ersetzt oder verändert werden. Beim Gen Editing werden durch verschiedene Techniken gezielt positionsspezifische DNA-Brüche erzeugt, an denen nicht immer Reparaturmechanismen beteiligt sind. Es besteht aus zwei Techniken – Inaktivierung und Korrektur.
Bei der Inaktivierung wird ein Zielgen ausgeschaltet, während die Korrektur die Reparatur des defekten Gens durch einen Bruch im Gen ermöglicht. Das Gen-Editing bietet ein enormes Potenzial für eine Vielzahl von Bereichen, darunter die Wirkstoffentwicklung, die Genchirurgie, Tiermodelle, die Untersuchung und Behandlung von Krankheiten, die Bereiche Lebensmittel, Biokraftstoffe und Biomaterial-Synthese und mehr.
Obwohl CRISPR als wichtige Gen-Editing-Technik derzeit ausgiebig genutzt wird, wurde das Gen-Editing selbst erstmals in den späten 1900er Jahren untersucht. Seit dem Aufkommen der CRISPR-Technik, einer vormals ehrgeizig verfolgten Anwendung, hat sich die Gentherapie zur gefragtesten Anwendung des Gen-Editings entwickelt. Diese kann durch zwei Ansätze umgesetzt werden: die Genaddition, bei der das vorhandene genetische Material ergänzt wird, um fehlerhafte oder fehlende Gene zu ersetzten, und das Gen-Editing, wobei Krankheiten durch die direkte Modifikation der krankheitsrelevanten DNA behandelt werden.
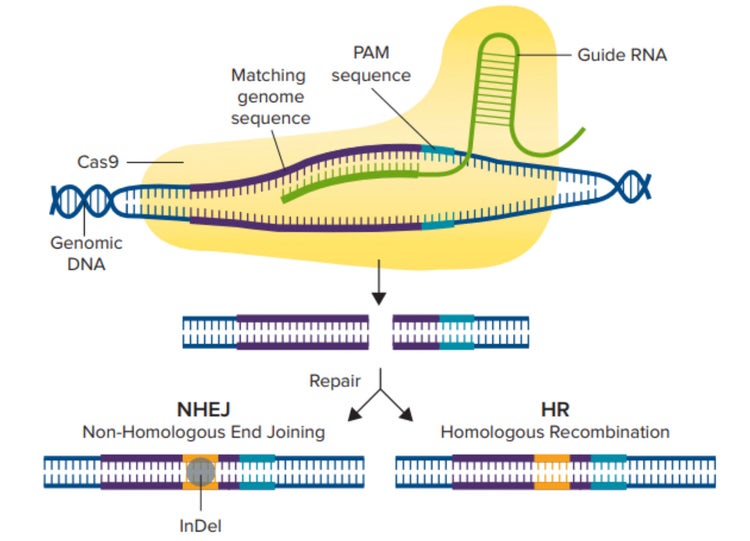
CRISPR/Cas9-Mechanismus. Das Cas9-Enzym wird aktiviert, indem es zunächst an eine Leit-RNA und dann an die passende genomische Sequenz bindet, die unmittelbar vor der aus 3 Nukleotiden bestehenden PAM-Sequenz liegt. Das Cas9-Enzym erzeugt dann einen Doppelstrangbruch, und entweder der NHEJ- oder der HDR-Weg wird genutzt, um die DNA zu reparieren, was zu einer editierten Gensequenz führt.
Eine Leit-RNA (gRNA, guide RNA), die einer crRNA (CRISPR-RNA) gleicht, ist so konzipiert, dass sie eine bestimmte Region innerhalb des Gens erkennt, und das Cas9-Enzym kann in dieser spezifischen Region des Wirtszell-Genoms Doppelstrangbrüche erzeugen (Abbildung 1). Nachdem ein Doppelstrangbruch erzeugt wurde, durchläuft die Zelle einen von zwei Reparaturwegen: den Weg der nicht-homologen Endverknüpfung (NHEJ, nonhomologous end joining) oder den Weg der Homologie-gerichteten Rekombination (HDR, homology-directed recombination). Der NHEJ-Weg wird üblicherweise durchlaufen, um Gene durch Insertionen oder Deletionen (Indels) von Basen zu zerstören. Der HDR-Weg kann hingegen genutzt werden, um durch den Austausch von Sequenzen zwischen zwei ähnlichen oder identischen DNA-Molekülen ein Reportergen oder eine editierte Gensequenz einzuschleusen.
Scale-up des Gen-Editings mit der CRISPR-Technik
„CRISPR – Clustered Regularly Interspaced Short Palindromic Repeats“ (zu Deutsch etwa: gehäuft auftretende, regelmäßig durch Spacer unterbrochene, kurze Palindrom-Wiederholungen). Diese DNA-Sequenzen wurden erstmals als Teil des Immunsystems von Prokaryoten wie Bakterien und Archaeen entdeckt und haben seit 2012 als Gen-Editier-Werkzeug an Bedeutung gewonnen (Jinek et al., 2012). Dies ist äußerst vielversprechend für eine Vielzahl von Anwendungen, darunter die Landwirtschaft, Krankheitsmodelle, Gentherapie und die Wirkstoffforschung, um nur einige zu nennen. Seine Präzision macht es zu einem perfekten Werkzeug für die Insertion (Knock-ins) und die Deletion (Knock-outs) sowie für andere Modifikationen von DNA-Sequenzen. Es hat die bisherigen mühsamen und teuren Gen-Editing-Werkzeuge wie TALENS (transcription activator-like effector nucleases, auf Deutsch: Transkriptionsaktivator-artige Effektor-Nukleasen) und ZFNS (Zinkfinger-Nukleasen) weitgehend ersetzt.
CRISPR-Sequenzen beinhalten nach jeder Palindrom-Wiederholung die DNA von früheren viralen Eindringlingen, die sogenannten Spacer, die bei der Erkennung und Zerstörung zukünftig eindringender ähnlicher Viren helfen. Das Verständnis dieses Mechanismus (Jinek et al., 2012) führte zum ersten Einsatz von CRISPR in eukaryotischen Zellen (Cong L., et al., 2013) und später in anderen Zelltypen sowie in Organismen anderer Taxonomie. Die CRISPR/Cas9-Systeme bestehen aus zwei Hauptkomponenten, die einen Ribonukleoprotein-Komplex bilden. Die erste Komponente, die Leit-RNA, bindet an eine komplementäre DNA-Sequenz im Genom. Die zweite Komponente, Cas9 aus Streptococcus pyogenes (SpCas9), führt einen Doppelstrangbruch an der Zielposition ein. Ein Protospacer-Nachbarmotiv PAM (protospacer adjacent motiif) ist der Ort, an dem die Nuklease zunächst bindet, damit an der stromaufwärts gelegenen Position ein Schnitt erfolgen kann. Verschiedene CRISPR-Nukleasen haben unterschiedliche PAM-Positionen, und sobald der Schnitt erfolgt ist, wird das Reparatursystem der Zelle aktiviert und es werden ebenfalls Modifikationen des Genoms eingeleitet.
Gen-Editing-Workflow
Der Workflow des Gen-Editings mit CRISPR-Mechanismen umfasst mehrere Schritte, um eine Zelllinie mit bestätigter Gen-Editierung zu erzeugen. Die effektive Optimierung dieser Schritte mit den richtigen Werkzeugen trägt zu einem effizienten Prozess bei, der die Zeit, den Aufwand und die Kosten verschiedener wissenschaftlicher Fortschritte verringert. Dieses Verfahren fördert die Beschleunigung der Forschung und Entwicklung und revolutioniert die Wirkstoffforschung, die Heilung von Krankheiten, die Produktion genetisch modifizierter Pflanzen usw. Um die wissenschaftliche Gemeinschaft weltweit bei der Verwirklichung ihrer Zielsetzungen mithilfe des Gen-Editings zu unterstützen, erörtern wir die notwendigen Schritte und die effektiven Lösungen, die wir hierzu anbieten.
Forschungslösungen für die Validierung von CRISPR/Cas9-vermittelten Gen-Edits
Die Instrumentenfamilie von Molecular Devices kann effektiv zur Durchführung/zum Screening von Experimenten eingesetzt werden, um den Erfolg von Gen-Editing-Projekten sicherzustellen. Der neue CloneSelect Imager Fluorescence (CSI-FL) liefert einen Nachweis der Monoklonalität am Tag 0 der Aussaat von Einzelzellen; zudem Daten zur Transfektionseffizienz, Zellkonfluenz und aus dem Mehrkanal-Fluoreszenz-Screening, um die Wirksamkeit des Gen-Editings bei kürzerer Nachverfolgungszeit zu validieren, ein geringes Risiko für den Transfer zu vieler Zellen und Robotik.
Zusätzlich kann unser SpectraMax i3x Multi-Mode Microplate Reader zur Bewertung der Transfektionseffizienz, zur Überwachung des Zellwachstums, zur Quantifizierung von DNA und Proteinen sowie zur Validierung von CRISPR/Cas9-vermittelten Gen-Edits mithilfe von ScanLater Western Blot-Analysen eingesetzt werden. Mit dem ImageXpress Micro Confocal System können qualitativ hochwertige Bilder von Autophagosomen aufgenommen werden, während die MetaXpress HCI Software einzelne Autophagosomen in jeder Zelle identifizieren und quantifizieren kann – so können Sie phänotypische Veränderungen analysieren, die aufgrund der CRISPR/Cas9-vermittelten Gen-Edits auftreten.
