QPix Microbial Colony Picker
Automatisiertes mikrobielles Screening-System mit der Fähigkeit, bis zu 3000 Kolonien pro Stunde zu picken
Automatisiertes mikrobielles Screening-System mit der Fähigkeit, bis zu 3.000 Kolonien pro Stunde zu picken
Der QPix® Microbial Colony Picker nutzt eine branchenführende Kolonie-Picking-Technologie, um Engpässe zu beseitigen und umfangreiche genetische Bibliotheken schnell, präzise und effizient zu screenen. Die einfach zu bedienende, intuitive Software führt die Benutzer durch die Konfiguration von Kolonie-Picking-Durchläufen, in denen Präzisionsroboter jedes Mal die richtigen Kolonien picken. Zusätzlich zum mikrobiellen Screening automatisiert das System mehrere Schritte der Probenvorbereitung und der Handhabung von Platten, wie z. B. den Transfer bakterieller Flüssigkulturen und das Ausplattieren auf Agar.
Die Daten werden automatisch in der Datenbank des Geräts aufgezeichnet, so dass den Benutzern ein vollständiger Audit Trail und eine Probennachverfolgung zur Verfügung stehen und niemals Daten verloren gehen. Unsere modulare, skalierbare Kolonie-Picker-Serie ermöglicht es Arbeitsgruppen jeglicher Größe, die Genauigkeit und den Durchsatz ihrer Arbeitsabläufe zu erhöhen und trotzdem auch zukünftig Durchsatzsteigerungen zu ermöglichen.

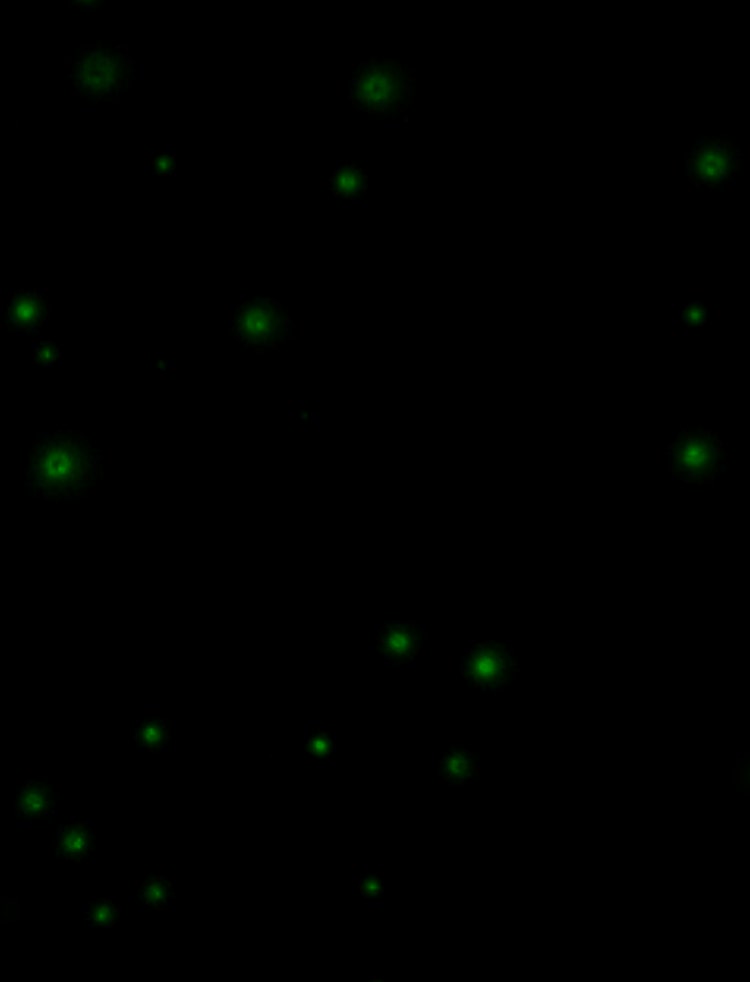
Identifizieren Sie Kolonien mit dem gewünschten Phänotyp
Die QPix Kolonie-Picker unterstützen eine große Vielfalt von Mikroorganismen und mehrere Auswahlmodi, darunter Fluoreszenzintensität, Blau/Weiß-Selektion, Größe und Nähe und Hemmzone.

Wählen Sie Klone effizient aus
Eine Reihe organismenspezifischer Nadel- und Agar-Sensoren gewährleisten effizientes Picken. Das System leistet eine Pickeffizienz von > 98 %, so dass Sie es mit gutem Gefühl unbeaufsichtigt lassen können.

Sterilität beibehalten
Es stehen eine ganze Reihe von Sterilitätsfunktionen zur Verfügung, einschließlich einer UV-Licht-Anwendung zur Desinfektion des Instrumenteninneren sowie Nadelreinigung und Halogentrocknung.
Merkmale

Organismusspezifische Nadeln
Nadeln zum Picken unterschiedlicher Formen und Flächen maximieren die Effizienz für E. coli, Phagen und Hefe. Plattierungsspezifische Nadeln gewährleisten eine gleichmäßige Verteilung von Flüssigkulturen auf Agar.

Mehrere Imagingverfahren
Durch die Verwendung von Weißlicht, Fluoreszenz und Farbe können Kolonien aufgrund von vordefinierten Parametern gepickt werden. Die Verwendung von Filtern ermöglicht Anwendungen wie das Blau/Weiß-Screening von Kolonien.

Ausplattieren und Verteilen
Automatisiertes Ausplattieren und Ausstreichen von 96 Proben kann in 30 Minuten durchgeführt werden, was eine längere Zeit ohne Beaufsichtigung ermöglicht.

Replikations-, Raster- und Hit-Picking
Die automatisierte Handhabung und Nachverfolgung der Platten optimiert die Verwaltung von Proben und nachfolgenden Assays. QPix Kolonie-Picker bieten flexible Fähigkeiten zur Plattenreplikation, zum Rastern und für das Hit-Picking.

Agar-Abtastung
Ein Ultraschall-Sensor für die Agarhöhe erkennt Höhenunterschiede, die durch unterschiedliche Abgabevolumina entstanden sind, und ermöglicht so eine maximale Pickeffizienz.

Skalierbare Automatisierungsoptionen*
Das Model QPix HT ist eine roboterkompatible Lösung mit einem modularen Deck. Das Team für Technische Lösungen mit fortschrittlichem Workflow kann einen Kolonie-Picker mit einer Vielfalt von benutzerdefinierten Serviceleistungen benutzerspezifisch anpassen.
*Preis, Lieferungszeit und Spezifikationen variieren aufgrund beiderseits abgesprochener technischer Anforderungen. Die Lösungsanforderungen können zu Anpassungen der Standardleistung führen.
Automatisieren Sie Ihren Arbeitsablauf mit dem QPix Colony Picker
Das Picken von Kolonien ist in der biologischen Forschung ein wesentlicher Schritt, da Forscher häufig mikrobielle Klone isolieren, um DNA oder Proteine in Massenproduktion herzustellen, damit diese downstream in einer Vielzahl von Anwendungen eingesetzt werden können. Traditionell wird das Picken von Kolonien manuell mit sterilen Pipettenspitzen oder Impfösen durchgeführt. Dies ist ein langsamer, arbeitsintensiver und zeitaufwendiger Prozess. Automatisierte Kolonie-Picker beschleunigen den ganzen Prozess nicht nur, die Ergebnisse sind zudem auch noch einheitlicher und zuverlässiger.
Welcher QPix ist der Richtige für Sie?
imaging
picking-capacity
colony-selection-criteria
picking-and-regional-picking
barcode-tracking
re-arraying-and-replication
gridding
plating-and-streaking
agar-to-agar
robotics-integration
shaking-incubator
liquid-handler
pcr
sealer-peeler
elisa
destination-plate-capacity
stackers
source-plate-capacity
walkaway-time
*Preis, Lieferungszeit und Spezifikationen variieren aufgrund beiderseits abgesprochener technischer Anforderungen. Die Lösungsanforderungen können zu Anpassungen der Standardleistung führen.
Benutzerdefinierte Lösungen unterliegen den Kaufbedingungen für benutzerdefinierte Produkte von Molecular Devices, erhältlich unter https://moleculardevices.com/custom-products-purchase-terms
Häufig gestellte Fragen
Was ist ein QPix Mikrobieller Kolonie-Picker?
Der QPix Mikrobielle Kolonie-Picker ist ein fortschrittliches automatisiertes System, das für die Selektion und das Picken von mikrobiellen Kolonien im Hochdurchsatzverfahren entwickelt wurde. Es rationalisiert den Prozess des Klon-Screenings und macht das manuelle Picken von Kolonien überflüssig.
Wie funktioniert der QPix Mikrobielle Kolonie-Picker?
Der QPix Mikrobielle Kolonie-Picker nutzt eine Kombination aus Imaging- und Robotertechnologie, um einzelne mikrobielle Kolonien genau zu identifizieren und von Agarplatten zu picken. Er verwendet hochentwickelte Algorithmen zur Analyse von Größe, Form und anderen Merkmalen der Kolonie und ermöglicht so eine präzise Kolonie-Selektion.
Was sind die wichtigsten Funktionen des QPix Mikrobiellen Kolonie-Pickers?
Der QPix Mikrobielle Kolonie-Picker bietet mehrere wichtige Funktionen, darunter hochauflösendes Imaging, anpassbare Kriterien für die Kolonieauswahl, automatisches Picken und Neuanordnung, einstellbare Picking-Parameter und intuitive Software für die Kolonieverfolgung und Datenanalyse.
Was sind die Vorteile des QPix Mikrobiellen Kolonie-Pickers?
Durch die Automatisierung des Kolonie-Pick-Prozesses erhöht der QPix Microbial Colony Picker den Durchsatz und die Effizienz beim Klon-Screening erheblich. Er minimiert das Risiko einer Kreuzkontamination, verbessert die Genauigkeit und Reproduzierbarkeit und setzt wertvolle Zeit und Ressourcen für die Forscher frei.
Welche Anwendungen können vom QPix Mikrobielle Kolonie-Picker profitieren?
Der QPix Mikrobielle Kolonie-Picker eignet sich ideal für eine Vielzahl von Anwendungen, darunter die rekombinante Proteinexpression, Antikörper-Discovery, synthetische Biologie, gerichtete Evolution und Stammentwicklung. Er eignet sich für die akademische Forschung, die pharmazeutische Entwicklung, die Biotechnologie und andere Branchen, die ein effizientes Klon-Screening benötigen.
Was sind die wesentlichen Funktionen von QPix Mikrobiellen Kolonie-Pickern?
QPix Mikrobielle Kolonie-Picker bieten automatisierte Fähigkeiten zum Picken, Identifizieren und Verfolgen von Kolonien. Sie ermöglichen es Forschern, einzelne mikrobielle Kolonien effizient von Agarplatten zu selektieren und zu übertragen, was Zeit spart und menschliche Fehler reduziert.
In welchen Arbeitsabläufen kann der QPix Mikrobielle Kolonie-Picker eingesetzt werden?
QPix Mikrobielle Kolonie-Picker sind vielseitige Werkzeuge, die in verschiedene Arbeitsabläufe integriert werden können. Sie werden häufig für Anwendungen wie Klon-Screening, Generierung von Mutantenbibliotheken, mikrobielle Genomik, gerichtete Evolution und Stammentwicklung mit hohem Durchsatz eingesetzt.
Wie optimieren QPix Mikrobielle Kolonie-Picker die wissenschaftliche Forschung?
QPix Mikrobielle Kolonie-Picker optimieren den Prozess des Klon-Screenings und des Kolonie-Pickens, sodass Forscher eine größere Anzahl an Kolonien in kürzerer Zeit analysieren können. Diese Optimierung verbessert die Effizienz von Forschungsprojekten, beschleunigt die Entdeckung und erleichtert Downstream-Analysen und -Experimente.
Können QPix Mikrobielle Kolonie-Picker verschiedene Arten von Agarplatten verarbeiten?
Ja, die QPix Mikrobielle Kolonie-Picker sind für verschiedene Agarplattenformate ausgelegt, darunter Petrischalen, Multiwell-Platten und andere benutzerdefinierte Platten. Das System ist flexibel und kann an die spezifischen Bedürfnisse verschiedener Experimente angepasst werden.
Wie verbessert die Begleitsoftware des QPix Mikrobiellen Kolonie-Pickers die Forschungsproduktivität?
Die mit den QPix Mikrobiellen Kolonie-Pickern bereitgestellte Software bietet intuitive Benutzeroberflächen für die Versuchsplanung, die Anpassung der Kriterien für die Kolonieselektion und die Datenanalyse. Sie ermöglicht eine effiziente Verfolgung von Kolonien, erleichtert das Workflow-Management und liefert aufschlussreiche Metriken für datengesteuerte Entscheidungen.
Neueste Ressourcen
Ausgewählte Anwendungen
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/biofuels
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/blue-white-screening
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/dna-sequencing
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/monoclonal-antibodies-mabs
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/phage-display
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/protein-evolution
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/synthetic-biology
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/plasmid-production
Bahnbrechende Kundenerfolge

ERFOLGSBERICHT
Inscripta ermöglicht Wissenschaftlern die Durchführung von digitalem Genomediting …
Wie können wir Sie dabei unterstützen, Ihre nächste große Entdeckung voranzubringen?
Unsere hochqualifizierten Teams unterstützen unsere Kunden an der Front, führen Produktdemonstrationen vor Ort oder per Fernzugriff durch und bieten Webinare und mehr, um Ihnen bei der Lösung Ihrer besonderen Forschungsherausforderungen zu helfen. Wie können wir Sie heute unterstützen?
Ich möchte …
Anwendungen für die QPix 400 Series Microbial Colony Picker
Technische Daten und Optionen für die QPix 400 Series Microbial Colony Picker
Ressourcen für die QPix 400 Series Microbial Colony Picker
Anzahl der Quellennachweise*: 371
Aktuellster Quellennachweis: Für eine vollständige Liste, klicken Sie hier.
*Quelle: https://scholar.google.com/