
MetaMorph Software zur Mikroskopie-Automatisierung und Bildanalyse [Eingestellt]
*BITTE LESEN* Benachrichtigung über Einstellung der Produktion
Molecular Devices evaluiert kontinuierlich Technologien zur Automatisierung von Mikroskopie und Bildanalyse, um sicherzustellen, dass ein Produkt die Erwartungen unserer Kunden erfüllt. Nach reiflicher Überlegung haben wir beschlossen, unser Angebot für die MetaMorph Software Suite, einschließlich MetaFluor und MetaVue, nach 7. Juli 2023 einzustellen. Diese Entscheidung entspricht unserer Strategie, neue innovative Produkte mit verbesserter Sicherheit, Design und Funktionalität zur Unterstützung der wissenschaftlichen Forschung anzubieten.
Mikroskop-Imaging-Software mit optionaler Analysesoftware für die Mikroskopie
Die MetaMorph® Software zur Mikroskopie-Automatisierung und Bildanalyse automatisiert die Erfassung, die Gerätesteuerung und die Bildanalyse. Sie integriert unterschiedliche Fluoreszenzmikroskop-Hardware und Peripheriegeräte ganz einfach in einer einzigen benutzerdefinierten Workstation. Die Software bietet viele benutzerfreundliche Anwendungsmodule für spezifische biologische Analysen. Das Portfolio umfasst zwei sich gegenseitig ergänzende Pakete: die MetaFluor® Software für Fluoreszenzverhältnis-Imaging und die MetaVue® Software für grundlegende Bilderfassung und -verarbeitung.

Unterstützt Mikroskope von Drittanbietern
Die MetaMorph Software funktioniert mit vielen gewerblich erhältlichen Mikroskopen, Laseranregungen und der TIRF-Optik, und kann auf bereits installierten, mit MetaMorph kompatiblen Imaging-Systemen aktiviert werden.

Messen Sie Fluoreszenzverhältnis-Imaging
Die MetaFluor®Fluoreszenzverhältnis-Bildgebungssoftware ist für intrazelluläre Ionenmessungen mit Einzel- oder Multiwellenlängen konzipiert. Sie ermöglicht so tiefere Einblicke in den Ionenaustausch und intrazelluläre Funktionen.

Dokumentieren und analysieren Sie Abbildungen
Das MetaVue™ Forschungs-Imaging-System ist eine einfache, benutzerfreundliche Softwareanwendung für die Erfassung und Verarbeitung von Bildern, die Ausführung von grafischen Funktionen sowie die Archivierung und den Abruf von Bildern.
Merkmale

Verarbeitung von Abbildungen in Echtzeit
Die Verarbeitung von Abbildungen wird durch die Hardwarebeschleunigung der Grafikbearbeitungseinheit unterstützt. Diese löst subzelluläre Objekte mit einer geringen Größe räumlich bis zu 20 nm und axial bis zu 40 nm auf.

Mehrdimensionales Aufnahmemodul
Ermöglicht mittels einer flexiblen, geführten Benutzeroberfläche die Erfassung komplexer Aufnahmesequenzen.
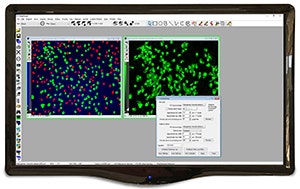
Intergierte morphometrische Analyse
Misst und kategorisiert Objekte in getrennte benutzerdefinierbare Klassen auf Grundlage einer beliebigen Kombination von morphometrischen Parametern, z. B. Form, Größe oder optische Dichte.

Scan-Objektträger-Modul
Automatisiert die Aufnahme mehrerer Bilder und setzt diese dann nahtlos zusammen. Diese ideale Lösung für große Gewebeproben stellt die Reproduzierbarkeit sicher und macht Spekulationen bei Tiling-Experimenten unnötig.

Geräte- und Kamera-Streaming
Beschleunigt die Bilderfassungsrate und überträgt während der Erfassung gleichzeitig Bilder auf den Speicher, wodurch dynamische zelluläre Ereignisse in Anwendungen wie kinetischem oder Lebendzell-Imaging erfasst werden.

4D-Viewer mit 3D-Messungen
Werkzeuge für mehrdimensionale Visualisierung, einschließlich Stapel sequenzieller Abbildungen, mehrere Z-Schnitte, Wellenlängen, Zeitpunkte und Positionen. Die Daten können für 3D-Isoflächenanzeige und -rotation gerendert werden.
Neueste Ressourcen
Ausgewählte Anwendungen
Bahnbrechende Kundenerfolge

ERFOLGSBERICHT
