
MetaXpress Software zur High-Content-Bilderfassung und -analyse
Multi-Level-Analysewerkzeuge für ein großes Spektrum an Anwendungen, die für ImageXpress Instrumente optimiert sind
Analysesoftware zur High-Content-Bilderfassung mit Zeitrafferanalyse-Funktion
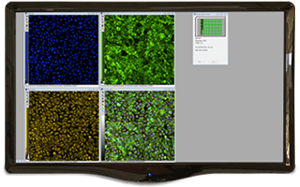
Die MetaXpress® Software zur High-Content-Bilderfassung und -analyse ist eine umfassende Lösung zur High-Content-Analyse, die einen streng organisierten und integrierten Arbeitsablauf bietet. Das Portfolio von Anwendungsmodulen erfüllt eine große Bandbreite von Bedürfnissen, angefangen bei einer einfachen Handhabung bis hin zum individuellen Assaydesign. Diese Software umfasst leistungsstarke und elegante Werkzeuge für das 2D- und 3D-Imaging, wie z. B. die Zeitrafferanalyse. Mit Unterstützung durch unseren Aufnahmeerfassungs-Konfigurationsassistenten werden Sie Daten in wenigen Minuten generieren.

Machen Sie Aufnahmen und Analysen in 2D und 3D
Die MetaXpress® Software ist eine Plattform für die 2D- und 3D-Bildaufnahme und -analyse. Das 3D-Bildanalyse-Modul kann Spheroide, Mikrogewebe, Zellen in einer 3D-Matrix und kleine Organismen analysieren.

Optimieren Sie den Bildanalyseprozess
Die modulare Toolbox ermöglicht die schnelle Konfiguration von Hunderten routinemäßig genutzter High-Content-Assays. Für größeren Komfort wählen Sie ein Modul aus unserem optionalen Angebot an schlüsselfertigen Anwendungsmodulen aus.

Gestalten Sie benutzerdefinierte Analysen mit Leichtigkeit
Erstellen Sie mehrschrittige Programme schnell mit dem benutzerdefinierten Modul-Editor. Er ermöglicht fortgeschrittene Analysen, wie z. B. Objekte innerhalb von Objekten zu identifizieren und morphometrische Klassifikatoren für die Formanalyse zu erstellen.
Merkmale

Z-Stapel und volumetrische 3D-Analyse
Die integrierten Aufnahmemodule für die Aufnahme und Analyse von 3D-Zellmodellen vereinfachen die Hoch-Durchsatz-Quantifizierung von 3D-Strukturen mit Volumen-, Intensitäts- und Distanzmessungen.

Schnelle Erfassung und Analyse
Erfasst schnell ablaufende biologische Funktionen ganz einfach mit unserer patentierten schnellen Aufnahmefrequenz für Serienbilder, die 50 Bilder pro Sekunde erfassen kann. Eine 1-Farben-, 384-Well-Platten-Analyse kann in nur sechs Minuten durchgeführt werden.

Intelligente Segmentierung
Die Anwendungsmodule bieten eine fertig voreingestellte Bildsegmentierung für labelfreie und fluoreszent markierte Abbildungen. Der benutzertedinierte Modul-Editor bietet fortgeschrittene Werkzeuge für eine verbesserte Segmentierung.

Skalierbares paralleles Prozessieren
Die MetaXpress PowerCore™ Software führt Analyseroutinen innerhalb von Minuten durch. Das optionale Modul nutzt die Leistung der Parallelprozessierung wirkungsvoll zur Beschleunigung von Analysen.

Datenverwaltungslösung
Der mit der MetaXpress® Software gelieferte MDCStore™ Datenmanager organisiert Bilder, experimentelle Informationen und die Datenanalyse. Er ermöglicht die nahtlose Verarbeitung von komplexen Bildsätzen und Metadaten.
Fortgeschrittene Informatik
Die AcuityXpress™ High-Content Informatik-Software ist eine optionale professionelle Plattform für fortgeschrittene Datenbankauswertung, Analyse, Statistik und Visualisierung. Für eine nahtlose Integration teilt sie sich die Datenbank mit MDCStore.
Schlüsselfertige Lösungen für die häufigsten Analyseroutinen
MetaXpress-Anwendungsmodule
Beschleunigen Sie mit der MetaXpress Software die Bildanalyse und die Datenverarbeitung – durch Anwendungsmodule, die Ihnen eine schnelle und einfache Datenanalyse erlauben:
- Mikrokerne
- Neuritenauswuchs
- Gefäßbildung Angiogenese
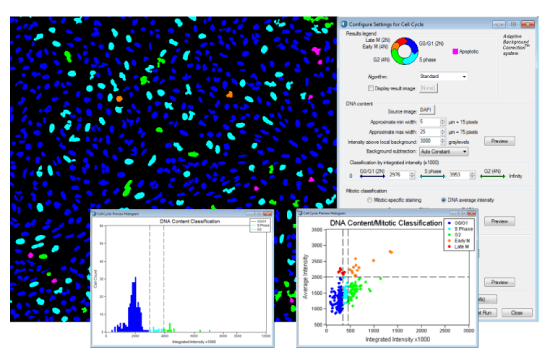
- Mitotischer Index
- Plus viele Anwendungen mehr …
Neueste Ressourcen
Ausgewählte Anwendungen
Bahnbrechende Kundenerfolge

ERFOLGSBERICHT
