Konstruktion von Next-Generation-Organoiden mit automatisierten Laborabläufen auf der #SLAS2022
Die SLAS2022, die Konferenz der „Society for Lab Automation and Screening“, bot eine weitere spannende Jahreskonferenz, bei der man sich über innovative Labortechnologien informieren konnte. Ganz gleich, ob Sie persönlich teilgenommen oder uns online auf unserer virtuellen Veranstaltungsseite besucht haben, wir freuen uns, Ihnen neue Methoden und Protokolle zur Automatisierung Ihrer komplexen Arbeitsabläufe in der Biologie vorstellen zu dürfen.
Hier ist ein kurzer Überblick über unsere Posterpräsentationen, die eine Vielzahl wertvoller Themen behandeln – von neuen Fortschritten in der Entwicklung von Organoiden der nächsten Generation bis hin zur Entwicklung eines automatisierten Labor-Arbeitsablaufs für 3D-Zellkulturen, Überwachung und High-Content-Imaging.
Wissenschaftliche Posterpräsentationen
1. Darm-Organoide für automatisierte Screening-Assays. High-Content-Imaging und -Analyse der Organoid-Morphologie
Autoren: Oksana Sirenko, PhD, Sr. Scientist; Krishna Macha, PhD, Research Scientist; Angeline Lim, PhD, Applications Scientist
https://share.vidyard.com/watch/Sd2VsnvWqs5m2gHkwnvZdg
3D-Zellmodelle, die verschiedene Gewebe repräsentieren, wurden erfolgreich verwendet, um komplexe biologische Effekte, Gewebearchitektur und Funktionalität zu erforschen. Die Komplexität von 3D-Modellen stellt jedoch nach wie vor eine Hürde für die breitere Akzeptanz in der Forschung und dem Wirkstoffscreening dar.
Hier beschreiben wir einen Arbeitsablauf zur Automatisierung der Organoidkultur. Die automatisierte Methode nutzt eine integrierte Arbeitszelle, die aus mehreren Instrumenten besteht, die eine automatisierte Zellkultur, Überwachung und High-Content-Imaging ermöglichen. Das integrierte System umfasst unser ImageXpress® Confocal HT.ai High-Content Imaging System, einen automatisierten CO2-Inkubator, einen automatisierten Liquid Handler (Biomek i7) sowie einen kollaborativen Roboter. Wir haben Methoden zur Automatisierung der Aussaat, des Medienaustauschs und der Überwachung der Entwicklung von Darmorganoiden entwickelt. Darüber hinaus ermöglicht die Methode die Automatisierung von Verbindungsprüfungen und die Bewertung phänotypischer Veränderungen.
Registrieren Sie sich für unsere Posterpräsentation, in der wir unsere Methoden besprechen und die Werkzeuge demonstrieren, die zur Erhöhung des Durchsatzes und zur Automatisierung von Organoid-Assays und des Screenings von Verbindungen verwendet werden. Wir werden auch Analyseansätze vorschlagen, die es Ihnen ermöglichen, mehr Informationen über diese komplexen Systeme, Krankheitsphänotypen und Verbindungseffekte zu gewinnen.
Registrieren Sie sich, um SLAS-Poster herunterzuladen
2. Automatisierung des Organ-on-a-Chip-Assays: Automatisiertes Kultivieren und Imaging sowie automatisierte Analyse der Angiogenese
Autoren: Angeline Lim, PhD, Applications Scientist; Oksana Sirenko, PhD, Sr. Scientist; Arthur Stok, Matthew Delport, Product Manager, MIMETAS; Francis Enane, Bhagya Wijayawardena
https://share.vidyard.com/watch/x5n3AMEEaf5xEAJCrC8jMW
Es werden dringend biologische Modellsysteme benötigt, die der menschlichen Biologie besser entsprechen. Dreidimensionale (3D) Zellmodelle und Organ-on-a-Chip-Strukturen, die verschiedene Gewebe repräsentieren, wurden erfolgreich zur Erforschung komplexer biologischer Effekte, Gewebearchitektur und Funktionalität eingesetzt. Das OrganoPlate® wurde als Organ-on-a-Chip-Plattform entwickelt, die die Bildung mikrofluidischer 3D-Langzeitkulturen von Lebendzellen ermöglicht. Die Komplexität von 3D-Modellen stellt jedoch nach wie vor eine Hürde für eine breitere Akzeptanz in der Forschung und dem Wirkstoffscreening dar. Die Automatisierung von Zellkulturen, Assays und Analysen kann die Werkzeuge bereitstellen, die zur Vereinfachung und Skalierung der Verwendung von Organ-on-a-Chip-Systemen erforderlich sind.
Hier beschreiben wir einen Arbeitsablauf zur Automatisierung von Organ-on-a-Chip-Kulturen sowie zur Überwachung und Automatisierung der Zellanalyse. Bei dieser automatisierten Methode wird eine integrierte Arbeitszelle verwendet, die aus mehreren Instrumenten besteht, die die Automatisierung und Überwachung von Zellkulturen ermöglichen. Wir haben Methoden zur Automatisierung von Zellaussaat, Medienaustausch und zur Überwachung der Entwicklung und des Wachstums von 3D-Gefäßen entwickelt. Darüber hinaus ermöglichen diese Methoden die automatisierte Prüfung von Verbindungen und die Beurteilung von Toxizitätseffekten. Wir haben den Angiogenese-Assay als Beispiel für den Automatisierungsprozess verwendet.
Melden Sie sich für unsere Posterpräsentation an, in der wir die phänotypischen Auslesungen überprüfen, die eine quantitative Beschreibung des Ausmaßes und der Komplexität der angiogenen Sprossen in 3D ermöglichten. Wir werden auch besprechen, wie die automatisierte Methode für das Screening von Verbindungen und die Verwendung der Organ-on-Chip-Technologie im High-Throughput-Format umfassend anwendbar ist.
Registrieren Sie sich, um SLAS-Poster herunterzuladen
3. Automatisierungsbasierter Arbeitsablauf für 3D-Organoidkulturen mit labelfreier Deep-Learning-Bildanalyse
Autoren: Angeline Lim, PhD, Applications Scientist; Misha Bashkurov, PhD, Product Owner; Joe Chen, Automation Solutions Engineer; Oksana Sirenko, PhD, Sr. Scientist
https://share.vidyard.com/watch/iaMAJtdpJD5Dq5VFoqSmg5
Das 3D-Zellkulturmodellsystem wird immer beliebter, da es die In-vivo-Mikroumgebung besser nachbildet als 2D-Zellkulturen. 3D-Organoide sind zelluläre Aggregate, die aus pluripotenten Stammzellen oder adulten Stammzellen gewonnen werden und sich selbst in organähnliche Strukturen organisieren können. Diese Organoide verfügen über die Kapazität für eine stabile Differenzierung und schnelles Wachstum, und als solches bietet das Organoid-Modellsystem ein großes Potenzial in der Krankheitsmodellierung, dem Wirkstoffscreening und der Präzisionstherapie.
Die zur Verwendung von Organoiden als Modellsystem verfügbaren Technologien befinden sich im Vergleich zu etablierteren 2D-Kultur- oder Tiermodellen noch in den Kinderschuhen. Um die Reproduzierbarkeit von Organoiden zwischen den Chargen anzugehen und den Prozess der Organoidkultur zu standardisieren und zu verbessern, ist eine weitere Entwicklung erforderlich. Darüber hinaus sind aktuelle Protokolle zur Erzeugung und Erhaltung von Organoiden komplex und zeitaufwendig und erfordern eine umfangreiche manuelle Handhabung.
Registrieren Sie sich für unsere Posterpräsentation, da wir die Machbarkeit der Verwendung einer automatisierten Arbeitszelle für die Kultur und Überwachung von 3D-Lungenorganoiden demonstrieren. Diese Ergebnisse bilden den grundlegenden Arbeitsablauf, der an andere 3D-Zellmodelle wie Darm-, Gehirn- oder von Patienten stammendes Gewebe angepasst werden kann, und ermöglichen das Scale-Up der Organoidproduktion für andere Downstream-Anwendungen.
Registrieren Sie sich, um SLAS-Poster herunterzuladen
4. Vereinfachter, benutzerfreundlicher, automatisierter Arbeitsablauf für das phänotypische Profiling auf Grundlage des Cell-Painting-Assays
Autoren: Angeline Lim, PhD, Applications Scientist; Michael Hayes, PhD, Beckman Coulter Life Sciences; Francis Enane, PhD, Sr. Applications Scientist, Beckman Coulter Life Sciences; David Egan, PhD, Mitbegründer und CEO, Core Life Analytics; Victor Wong, PhD, Application Scientist, Core Life Analytics
https://share.vidyard.com/watch/EedQwmnwXDix1BCpDxYMVT
Multiparametrische High-Content-Screening-Ansätze, wie der Cell-Painting-Assay, werden zunehmend in vielen Anwendungen eingesetzt, von Wirkstoffforschungsprogrammen bis hin zum Screening der funktionellen Genomik. Der Cell Painting Assay verwendet bis zu sechs Fluoreszenzfarbstoffe, um eine Vielzahl von Organellen auf Einzelzellebene zu markieren und zu visualisieren. Morphologische Merkmale, die aus dem Assay extrahiert wurden, liefern einzigartige zelluläre „Signaturen“, die einen Überblick über die Zelle geben.
Zell-Painting-Assays werden üblicherweise im großen Maßstab mit mehreren Assayplatten durchgeführt. Die Arbeitsabläufe können zeit- und arbeitsintensiv sein und das Ausfüllen eines Bildschirms kann mehrere Tage in Anspruch nehmen. Der Einsatz von Automatisierung mit Pipettierrobotern könnte dazu beitragen, diese Prozesse zu optimieren, wertvolle Zeit zu sparen und den Assay-Durchsatz zu erhöhen. Darüber hinaus erfordert das schiere Datenvolumen, das aus diesen Experimenten generiert wird, leistungsstarke Software und Computerwerkzeuge, um aussagekräftige Informationen zu extrahieren. Die Rechenanforderungen, die für die Analyse dieser großen Datensätze erforderlich sind, können über die technischen Mittel kleinerer Forschungslabore hinausgehen.
Hier haben wir einen vollautomatischen Arbeitsablauf für den Cell-Painting-Assay mit den zusätzlichen Vorteilen von weniger Zeitaufwand und Fehlern bei der Handhabung durch den Anwender bei erhöhtem Assay-Durchsatz entwickelt.
Registrieren Sie sich, um SLAS-Poster herunterzuladen
5. Beurteilung der Thrombinerzeugung mit dem TECHNOTHROMBIN TGA-Kit auf dem SpectraMax i3x Multi-Mode Mikroplatten-Reader
Autoren: Cathy Olsen, PhD, Application Scientist
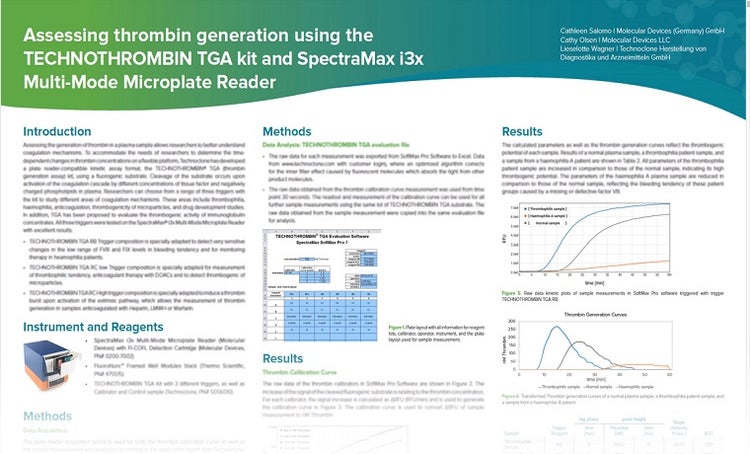
Die Beurteilung der Thrombinerzeugung in einer Plasmaprobe ermöglicht es, Koagulationsmechanismen besser zu verstehen. Um die Anforderungen von Forschern zur Bestimmung der zeitabhängigen Veränderungen der Thrombinkonzentrationen auf einer flexiblen Plattform zu erfüllen, hat Technoclone ein Plattenreader-kompatibles kinetisches Assay-Format entwickelt, das TECHNOTHROMBIN® TGA-Kit, das ein fluorogenes Substrat verwendet. Die Spaltung des Substrats erfolgt nach Aktivierung der Koagulationskaskade durch unterschiedliche Konzentrationen des Gewebefaktors und negativ geladener Phospholipide im Plasma.
Forscher können aus einer Reihe von drei Auslösern mit dem Kit auswählen, um verschiedene Bereiche der Koagulationsmechanismen zu untersuchen. Diese Bereiche umfassen Thrombophilie, Hämophilie, Antikoagulation, Thrombogenität von Mikropartikeln und Studien zur Wirkstoffentwicklung. Alle drei Trigger wurden mit dem SpectraMax® i3x Multi-Mode Mikroplatten-Reader mit ausgezeichneten Ergebnissen getestet.
Nach der Generierung der Daten in der SoftMax Pro® Software wurden die Daten in die TECHNOTHROMBIN TGA-Evaluationsdatei von Technoclone übertragen, um alle Parameter der Thrombinerzeugung auf Grundlage der Thrombinerzeugungskurve zu berechnen. Dieses Poster zeigt, dass die Kombination des SpectraMax i3x Readers und des TECHNOTHROMBIN TGA-Kits eine ideale Plattform für die Durchführung von Thrombinerzeugungs-Assays mit guter Präzision für Forschungszwecke bietet.
Registrieren Sie sich, um SLAS-Poster herunterzuladen
Entdecken Sie Lösungen zur Automatisierung Ihrer Arbeitsabläufe in der 3D-Biologie
Ganz gleich, ob Sie gerade erst damit beginnen, die Vorteile von 3D-Gewebemodellen und Imaging zu erforschen oder fortschrittliche 3D-Arbeitsabläufe zu nutzen – wir bieten Lösungen für die Komplexität, die mit der Erfassung und Analyse von Organoiden einhergehen.
Besuchen Sie unser Organoid Innovation Center und entdecken Sie unseren neuesten Raum für die Zusammenarbeit, in dem Kunden, Forscher und interne Wissenschaftler zusammenkommen, um automatisierte Arbeitsabläufe für die Organoidkultivierung und das Screening zu testen. Automatisieren Sie Ihre Arbeitsabläufe in der 3D-Kultur und übernehmen Sie schnell innovative biologische 3D-Methoden und -Technologien, um die Laborkosten zu senken und die Grenzen Ihrer Forschung zu erweitern.