
Zellzählsystem
Beim Zellzähler: HepG2-Zellen
HepG2 ist eine Zellinie, die aus dem Lebergewebe eines Patienten mit einem Leberzellkarzinom (HCC, hepatocellular carcinoma) gewonnen wurde. Sie wird häufig als ein Modellsystem für HCC sowie zur Untersuchung der Verstoffwechselung von Wirkstoffen und der Toxizität herangezogen. Die Kulturen sind adhärent mit einer epithelialen Morphologie und tendieren dazu, in kleinen Aggregaten zu wachsen. Dies macht das Auszählen einzelner Zellen schwierig. Unter den richtigen Kultubedingungen können sie deutlich unterscheidbare apikale und basale Oberflächen bilden, die für die Erforschung von Lebererkrankungen zugänglich sind.
Sehen Sie sich das Webinar zur StainFree-Zellerkennung an
StainFree Cell Detection Application Note herunterladen
StainFree-Zellerkennung Application Note herunterladen (China)
E-Book herunterladen: Zellen zählen wie ein Profi
Abbildung 1: Bestimmung des prozentualen bedeckten Bereichs

HepG2-Zellen wurden mit dem SpectraMax MiniMax 300 Imaging Cytometer aufgenommen und die Zellen wurden mit einer benutzerdefinierten Analyseeinstellung identifiziert. Auf der linken Seite ist das originale Durchlichtbild gezeigt. Auf der rechten Seite ist dasselbe Bild mit einer violetten Maske dargestellt, die die von der Software identifizierten Zellen hervorhebt. Zwei Zelldichten werden zum Vergleich dargestellt.
Abbildung 2: StainFree- vs. Fluoreszenzanalyse des prozentualen bedeckten Bereichs
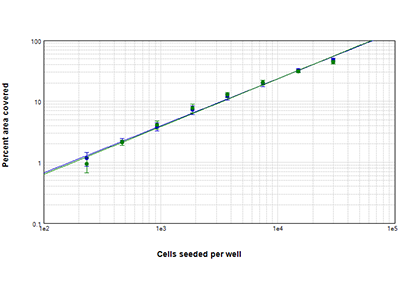
Die Konfluenz von HepG2-Zellen, die bei Zelldichten von 234 bis 30.000 Zellen pro Well ausgesät wurden, wurden mit der StainFree-Technologie (blaue Punkte) analysiert oder mit dem Farbstoff des EarlyTox™ Live Cell Assays gefärbt und die fluoreszenten Bereiche wurden analysiert. Der prozentuale bedeckte Bereich eines untersuchten Bildausschnitts stimmt für beide Methoden über alle Zelldichten hinweg nahezu überein.
Abbildung 3: EarlyTox Caspase-3/7 NucView 488 Assay
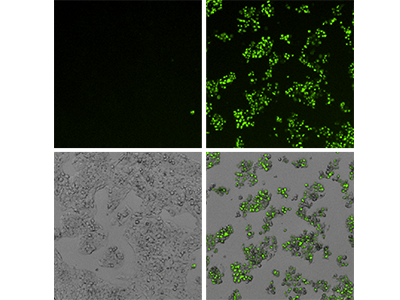
HepG2_Zellen, behandelt mit Kontrollmedium (linke Panels) oder 200 µM Capsaicin (rechte Panels), wurden mit dem EarlyTox Caspase-3/7 NucView 488 Assay-Kit untersucht und mit den Kanälen für grüne Fluoreszenz und Durchlicht (TL, transmitted light) des SpectraMax MiniMax 300 Imaging Cytometers aufgenommen. Obere Reihe: Fluoreszenzabbildung, die apoptotische Zellen zeigt, die mit grüner Fluoreszenz markiert wurden. Untere Reihe: Die überlagerten Durchlicht- und Fluoreszenzabbildungen zeigen eine minimale Apoptose in den Kontrollzellen und fast 100 % Apoptose in den behandelten Zellen.
Abbildung 4: EarlyTox Caspase-3/7 NucView 488 Assay (Zeitverlauf)
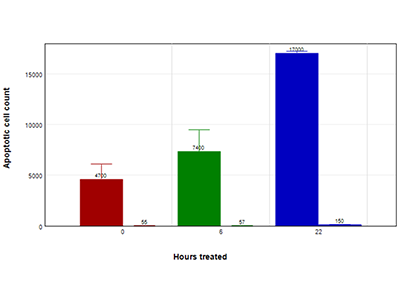
HepG2-Zellen, die über 4,5 Stunden (rote Balken), 6 Stunden (grüne Balken) oder 22 Stunden (blaue Balken) mit 200 µM Capsaicin (Balken links) oder Kontrollmedium (Balken rechts) behandelt wurden. Die Apoptose wurde mit dem EarlyTox Caspase-3/7 NucView 488 Assay-Kit bestimmt. Die Zellen wurden mit 5 µM des NucView 488-Substrats behandelt und zu den angegebenen Zeitpunkten aufgenommen.
Abbildung 5: EarlyTox Caspase-3/7 R110 Assay-Kit und EarlyTox Glutathione Assay-Kit
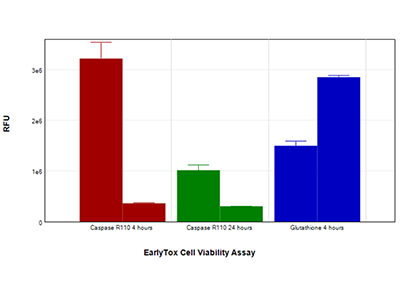
HepG2-Zellen, behandelt mit 200 µM Capsaicin (linker Balken jedes Paares) oder Kontrollmedium (rechter Balken jedes Paares), wurden mit dem EarlyTox Caspase-3/7 R110 Assay-Kit (rote und grüne Balken) untersucht, der die Caspase-Aktivität misst, oder dem EarlyTox Glutathione Assay-Kit (blaue Balken), der die Abnahme des Glutathions, einem frühen Indikator der Apoptose, misst. Beide Assays wurden auf dem SpectraMax i3x Multi-Mode Microplate Reader unter Verwendung vorkonfigurierter Protokolle der SoftMax Pro Software untersucht.
Tipp: HepG2-Zellen wachsen in Aggregaten oder Klumpen unregelmäßig geformter Zellen, die mit dem Auge nicht unterscheidbar sind. Die StainFree™-Technologie bietet eine hilfreiche Vorgehensweise zur Quantifizierung des HepG2-Zellwachstums oder der Konfluenz, um die mit Zellen bedeckten Bereiche zu bestimmen. Bilder, die im Durchlichtkanal aufgenommen wurden, können mit den „Field Analysis“-Einstellungen einfach analysiert werden. Es kann die vordefinierte „Cells“-Analyseeinstellung angewendet werden oder es kann mit den Zeichenwerkzeugen der SoftMax Pro Software eine benutzerdefinierte Analyse konfiguriert werden.
HepG2 Zellanalyse-Toolkit
- SpectraMax ® i3 Multi-Mode Mikroplatten-Detektionsplattform
- SpectraMax ® MiniMax™ 300 Imaging Cytometer
- SoftMax ® Pro Software
Instrumenteinstellungen
Art der Analyse: Field Analysis
Wellenlänge zum Auffinden von Objekten: TL
Über die Technologie der StainFree-Zellerkennung
Das Imaging zellbasierter Assays benötigt üblicherweise die Verwendung von fluoreszenten Sonden, die für lebende Zellen toxisch sein können oder möglicherweise nur in fixierten Zellen funktionieren. Eine labelfreie Methode zur Analyse von Zellzahl und der Zellkonfluenz ermöglicht es Forschern, die Zellproliferation und Zellgesundheit quantitativ zu überwachen – ohne zeitaufwendige Arbeitsabläufe, die die Zellviabilität beeinträchtigen.
Die SpectraMax i3 Multi-Mode Microplate Platform mit dem MiniMax 300 Imaging Cytometer nutzt die zum Patent angemeldete StainFree-Zellbestimmungs-Technologie. Diese ermöglicht Ihnen, Zellproliferation-, Zytotoxizitäts- und andere Assays ohne Zellkernfärbungen wie DAPI, die sich in die DNA einfügen, oder Lebendzellfärbungen durchzuführen, die langfristig für Zellen toxisch sind.