
Zellzählsystem
Beim Zellzähler: PC-12-Zellen
Ursprünglich stammen PC-12-Zellen aus einem Phäochromozytom (neuroendokriner Tumor) aus dem Nebennierenmark der Ratte und wurden 1976 von Greene und Tischler kultiviert. Sie wurden als eine Modellzelllinie und als Alternative für primäre Zellkulturen aus chromaffinen Zellen der Nebenniere entwickelt. PC-12-Zellen besitzen die Fähigkeit, sich in der Anwesenheit des Nervenwachstumsfaktors (nerve growth factor) oder Dexamethason zu Zellen zu differenzieren, die Nervenzellen ähneln. Wegen ihrer Differenzierungsfähigkeit und der einfachen Kultivierung werden PC-12-Zellen in vielen verschiedenen Forschungsgebieten verwendet, angefangen bei der Erforschung von Wirkstoff-Wirkungen bis hin zur Neurosekretion.
Sehen Sie sich das Webinar zur StainFree-Zellerkennung an
StainFree Cell Detection Application Note herunterladen
E-Book herunterladen: Zellen zählen wie ein Profi
Abbildung 1. StainFree-Technologie im Vergleich zur Zellzahlbestimmung mittels Fluoreszenz
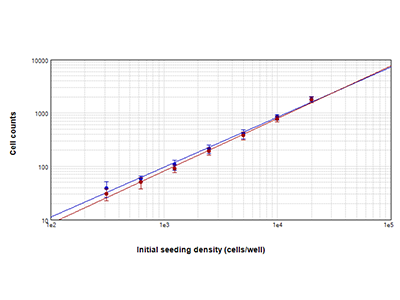
PC-12-Zellen, die mit der StainFree™-Zellbestimmungs-Technologie (blaue Kreise) und roter Zellkernfärbung (rote Kreise) ausgezählt wurden. Die Zellzahlen aus beiden Methoden stimmen fast genau überein. Dies beweist, dass die StainFree-Technologie genaue Zellzahlbestimmungen liefert und gleichzeitig die Notwendigkeit für Fluoreszenzfarbstoffe beseitigt (R2 > 0,99 für jeden Graph).
Abbildung 2. Wachstumskurven, die aus StainFree-Analysen der zellbedeckten Bereiche erhalten wurden
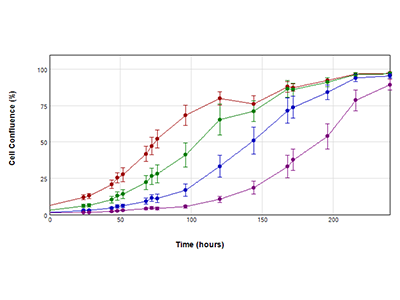
Wachstumskurven von PC-12-Zellen über einen Zehn-Tages-Zeitraum. Die Zellen wurden anfangs bei 2000 Zellen (rot), 1000 Zellen (grün), 500 Zellen (blau), und 200 Zellen (violett) ausgesät. Alle 24 Stunden wurde die Zellkonfluenz mit der Funktion „Field Analysis“ der SoftMax Pro Software gemessen. Der Abfall der Konfluenz nach 144 Stunden (*), der in der mit einer Ausgangsdichte von 2000 Zellen ausgesäten Population beobachtet wurde, ist wahrscheinlich durch einen Mangel an Nährstoffen in der Kultur verursacht. Ursprünglich wurde das Medium alle drei Tage gewechselt, dann jedoch ab 144 Stunden täglich ausgetauscht.
Abbildung 3. StainFree-Analyse von PC-12-Zellen mit Mitochondrienfärbung
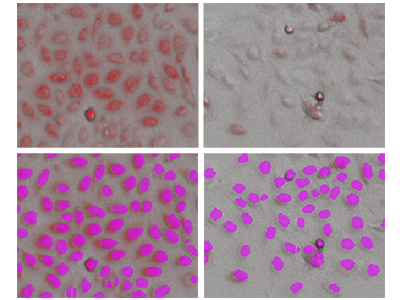
Die obere Reihe zeigt Durchlicht-Aufnahmen, die mit rot fluoreszierenden (Mitochondrien-)Aufnahmen überlagert wurden. Die untere Reihe zeigt eine StainFree-Analyse mit einzelnen Zellen (violette Maske), die von der Software identifiziert wurden. Links: unbehandelte Zellen, rechts: mit 1 µM Valinomycin behandelte Zellen. Für jede identifizierte Einzelzelle konnte die Intensität der Mitochondrienfärbung berechnet werden.
Abbildung 4. IC50-Kurven für Valinomycin-behandelte PC-12-Zellen
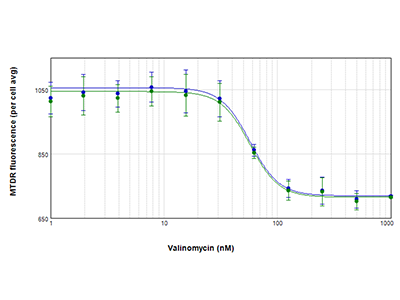
PC-12-Zellen wurden mit Valinomycin behandelt und mit dem Farbstoff MitoTracker™ Deep Red FM (MTDR) auf mitochondriale Aktivität untersucht. Gezeigt sind konzentrationsabhängige Reaktionskurven für Zellen, die mittels StainFree-Technologie (blau) oder fluoreszenter Zellkernfärbung (grün) analysiert wurden, um einzelne Zellen zu identifizieren. Die durchschnittliche mitochondriale Fluoreszenz pro Zelle ist verglichen mit der Konzentration von Valinomycin grafisch dargestellt. Mit beiden Analysemethoden wurden fast identische Kurven erzielt, und der IC50-Wert lag für beide Kurven bei 57 nm.
Tipp 1:
Um die PC-12-Zellen ohne Färbung auszuzählen empfehlen wir, mithilfe der Zeichenwerkzeuge eine neue Einstellung in der Software zu definieren. Wählen Sie die „Discrete Object Analysis“ und verwenden Sie dann die Zeichenwerkzeuge, um in Ihren maximal und minimal vergrößerten Aufnahmen Zellen und zellfreie Bereiche zu definieren. Um in dichten Anhäufungen von Zellen genauere Zellzahlen zu bestimmen, versuchen Sie, knapp innerhalb der Grenzen der Zellen zu zeichnen. Umgekehrt, wenn Sie feststellen, dass die Software in Ihren Aufnahmen zu viele Zellen zählt, versuchen Sie knapp außerhalb der Zellgrenzen zu zeichnen.
Tipp 2:
Es ist ganz einfach, mit der „Field Analysis“-Funktion der SoftMax Pro Software Wachstumskurven zu erstellen. Die Field Analysis berechnet den prozentualen Bereich, der in der Abbildung mit Zellen bedeckt ist (Konfluenz). Die Messung der Zellkonfluenz kann für die Entwicklung von Assays ein wertvolles Werkzeug sein. Sie können zum Beispiel Zellen bei zwei verschiedenen Test-Dichten aussäen, die Konfluenz ihrer Zellen kurz vor dem Assay berechnen und nach dem Erhalt der Assay-Ergebnisse bestimmen, welche Konfluenzstärke die besten Ergebnisse liefert.
PC-12 Zellanalyse-Toolkit
- SpectraMax® i3 Multi-Mode Microplate Detection Platform
- SpectraMax® MiniMax™ 300 Imaging Cytometer
- SoftMax® Pro Software
Instrumenteinstellungen
Über die Technologie der StainFree-Zellerkennung
Das Imaging zellbasierter Assays benötigt üblicherweise die Verwendung von fluoreszenten Sonden, die für lebende Zellen toxisch sein können oder möglicherweise nur in fixierten Zellen funktionieren. Eine labelfreie Methode zur Analyse von Zellzahl und der Zellkonfluenz ermöglicht es Forschern, die Zellproliferation und Zellgesundheit quantitativ zu überwachen – ohne zeitaufwendige Arbeitsabläufe, die die Zellviabilität beeinträchtigen.
Die SpectraMax i3 Multi-Mode Microplate Platform mit dem MiniMax 300 Imaging Cytometer nutzt die zum Patent angemeldete StainFree-Zellbestimmungs-Technologie. Diese ermöglicht Ihnen, Zellproliferation-, Zytotoxizitäts- und andere Assays ohne Zellkernfärbungen wie DAPI, die sich in die DNA einfügen, oder Lebendzellfärbungen durchzuführen, die langfristig für Zellen toxisch sind.